Els van den Brink
Spatial transcriptomics maakt het mogelijk om te meten welke genen worden afgelezen op elke locatie in een weefsel. Een fascinerende technologie in een verrassend groot aantal varianten.
Els van den Brink
Terwijl alle cellen in ons menselijk lichaam hetzelfde DNA bevatten, zijn ze toch heel verschillend. Een bloedcel, een hersencel en een huidcel zijn nauwelijks met elkaar te vergelijken, ondanks dat ze allemaal hetzelfde genoom in zich hebben. Dat heeft te maken met de mate waarin de verschillende genen worden afgelezen en vertaald naar RNA – ook wel het transcriptoom genoemd. In een bloedcel is het transcriptoom heel anders dan in een huidcel of hersencel. Vandaar dat steeds meer onderzoekers niet alleen het genoom van cellen analyseren, maar ook het transcriptoom. De laatste jaren was dit ook steeds beter mogelijk voor individuele cellen. Dit staat bekend als single cell transcriptomics.
Het gezegde ‘Hoe meer je weet, hoe meer je weet dat je niks weet’ is een uitspraak die op dit gebied ook zeker van toepassing is. Het is mogelijk allerlei cellen te karakteriseren, maar als niet precies bekend is wat hun locatie en hun omgeving is, dan kunnen onderzoekers er nog steeds niet zo veel mee. Bij immunotherapie tegen kanker, willen die niet alleen weten welke immuuncellen aanwezig zijn, maar ook waar ze precies gelokaliseerd zijn, en in hoeverre ze ook echt een tumor binnendringen.
“Spatial transcriptomics is als een vruchtentaart”
Vandaar dat er nieuwe technologieën zijn ontwikkeld die nog een stap verder gaan en de analyse van het transcriptoom kunnen linken aan de locatie van deze cellen in het weefsel, de zogenaamde spatial transcriptomics.
De oorspronkelijke transcriptomics-analyse is vergelijkbaar met het meten aan een smoothie, waarbij de resultaten een mix zijn van alle ingrediënten. Single cell transcriptomics ging al een stap verder, eerder een fruitsalade, met alle ingrediënten duidelijk herkenbaar, maar nog wel door elkaar gehusseld. Spatial transcriptomics, gaat nóg een stap verder en is als een vruchtentaart, waar alle verschillende fruitsoorten mooi op gerangschikt liggen. Zo is exact vast te te stellen waar welke soort ligt en hoe ze met elkaar verbonden zijn.
Spatial transcriptomics werd door Nature in 2020 uitgeroepen tot methode van het jaar. De technologie wordt vooral gebruikt voor wetenschappelijk onderzoek, met name op het gebied van immuno-oncologie en neurologie. Onderzoekers gebruiken het bijvoorbeeld voor onderzoek naar immunotherapie tegen kanker, en om de hersenen in kaart te brengen. In principe zou de technologie ook gebruikt kunnen worden voor diagnostisch onderzoek, in het kader van personalized medicine. Elke tumor heeft namelijk een unieke omgeving die bepaalde immuuncellen kan onderdrukken en de behandeling kan er veel baat bij hebben om dat voor elke individuele tumor apart vast te stellen.
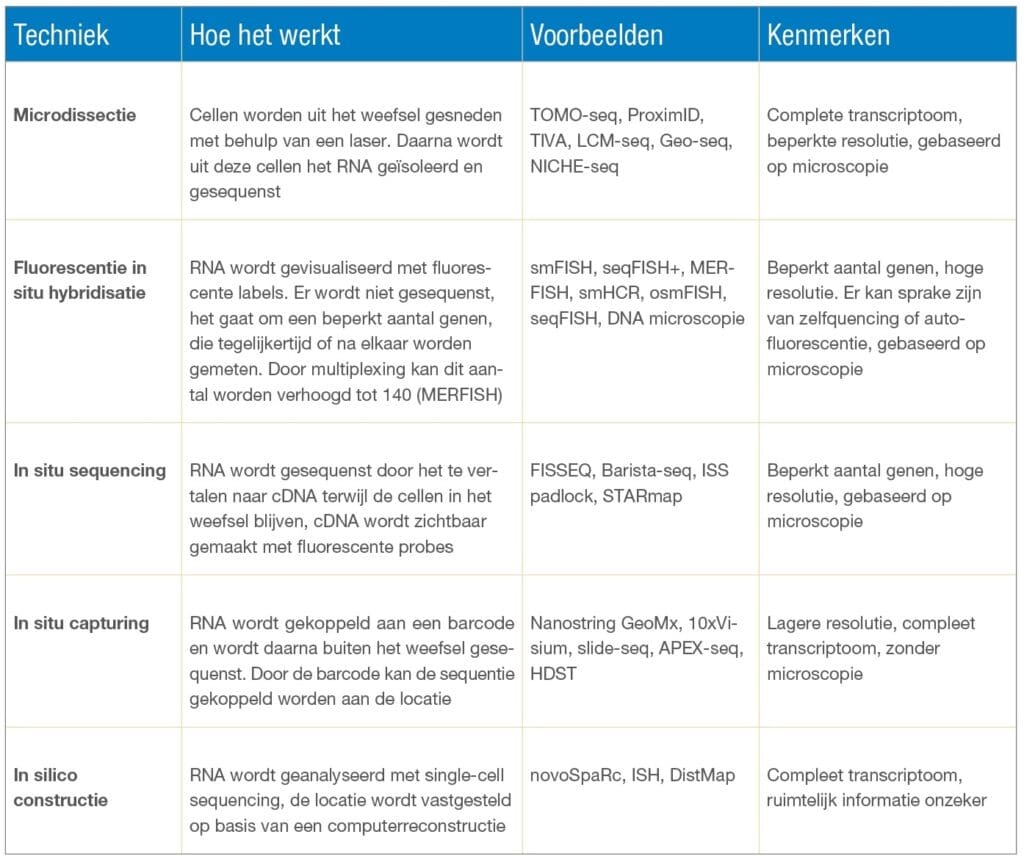
Spatial transcriptomics bestaat ondertussen in enorm veel verschillende varianten. Het zijn er zelfs zo veel, dat het overweldigend kan aanvoelen, al zitten er ook een aantal tussen die alleen worden toegepast binnen het instituut dat ze heeft ontwikkeld. Elke variant heeft weer net iets andere kenmerken en voor- en nadelen, bijvoorbeeld qua ruimtelijke resolutie, het aantal genen dat gemeten kan worden en de throughtput.
“Qua data-analyse zijn al snel de grenzen bereikt van wat haalbaar is”
Bij sommige technieken is de resolutie zo hoog, dat het mogelijk is om individuele cellen te onderscheiden, en soms zelfs subcellulaire delen zoals de celkern of organellen. Hierbij is het vaak wel zo dat het aantal genen dat gemeten kan worden beperkt is. Andere technieken hebben een minder hoge resolutie en kunnen naastgelegen cellen niet altijd van elkaar onderscheiden, maar hebben daarbij vaak wel weer de mogelijkheid om het complete transcriptoom te analyseren.
De varianten zijn in principe onder te verdelen in vijf groepen:
• microdissectie
• fluorescente in situ hybridisatie
• in situ sequencing
• in situ capturing
• in silico constructie (zie onderstaande tabel).
De kunst is om tussen al die varianten degene te vinden die het beste aansluit bij de eigen onderzoeksvraag. Malte Kühnemund, directeur R&D van 10x Genomics, een van de grootste spelers op het gebied van spatial transcriptomics, waarschuwt in een interview met labiotech.eu: ‘Het risico bestaat dat mensen gewoon maar eentje kiezen waarvoor de meeste reclame wordt gemaakt, zich dan realiseren dat ze niet de antwoorden krijgen die ze nodig hebben en het dan maar opgeven.’
“Gewoon maar eentje kiezen waarvoor de meeste reclame wordt gemaakt?”Malte Kühnemund, directeur R&D van 10x Genomics
Naast de verdere ontwikkeling van de technologie zelf is de verwerking van de data een van de grote uitdagingen. Door de combinatie van genetische en ruimtelijke informatie levert dit soort analyses al gauw enorme hoeveelheden data op, waarmee al snel de grenzen bereikt worden van wat haalbaar is qua data-analyse. Waarschijnlijk zal de data-analyse voorlopig de bottleneck zijn die bepaalt wat deze technieken aan waardevolle informatie kunnen opleveren.
• microdissectie
• fluorescente in situ hybridisatie
• in situ sequencing
• in situ capturing
• in silico constructie